如何用gggenes畫基因結(jié)構(gòu)圖
本篇文章為大家展示了如何用gggenes 畫基因結(jié)構(gòu)圖,內(nèi)容簡明扼要并且容易理解,絕對能使你眼前一亮,通過這篇文章的詳細介紹希望你能有所收獲。
讓客戶滿意是我們工作的目標,不斷超越客戶的期望值來自于我們對這個行業(yè)的熱愛。我們立志把好的技術(shù)通過有效、簡單的方式提供給客戶,將通過不懈努力成為客戶在信息化領(lǐng)域值得信任、有價值的長期合作伙伴,公司提供的服務項目有:申請域名、雅安服務器托管、營銷軟件、網(wǎng)站建設、連平網(wǎng)站維護、網(wǎng)站推廣。

install.packages("gggenes") 或者從github下載安裝:
devtools::install_github("wilkox/gggenes") 2. 準備輸入數(shù)據(jù)
官方給的例子如下:
> head(example_genes) molecule gene start end strand direction1 Genome5 genA 405113 407035 forward 12 Genome5 genB 407035 407916 forward 13 Genome5 genC 407927 408394 forward 14 Genome5 genD 408387 408737 reverse -15 Genome5 genE 408751 409830 forward 16 Genome5 genF 409836 410315 forward 1
輸入數(shù)據(jù)應該包含6列,分別代表:
物種名 基因名 起始位置 結(jié)束位置 基因方向 基因方向
如果不需要考慮畫出基因方向的話,只需要前4列數(shù)據(jù)就行:物種名,基因名,起始位置,結(jié)束位置
如果加上基因方向,就需要加上 strand 這一列,正負鏈分用“forward”和“reverse”表示。示例數(shù)據(jù)中的 direction 這一列是多余的,并不會被用到。
2. 作圖
library(ggplot2)library(gggenes)ggplot(example_genes, aes(xmin = start, xmax = end,y = molecule, fill = gene)) +geom_gene_arrow() +facet_wrap(~ molecule, scales = "free", ncol = 1) +scale_fill_brewer(palette = "Set3") +theme_genes()
圖如下:
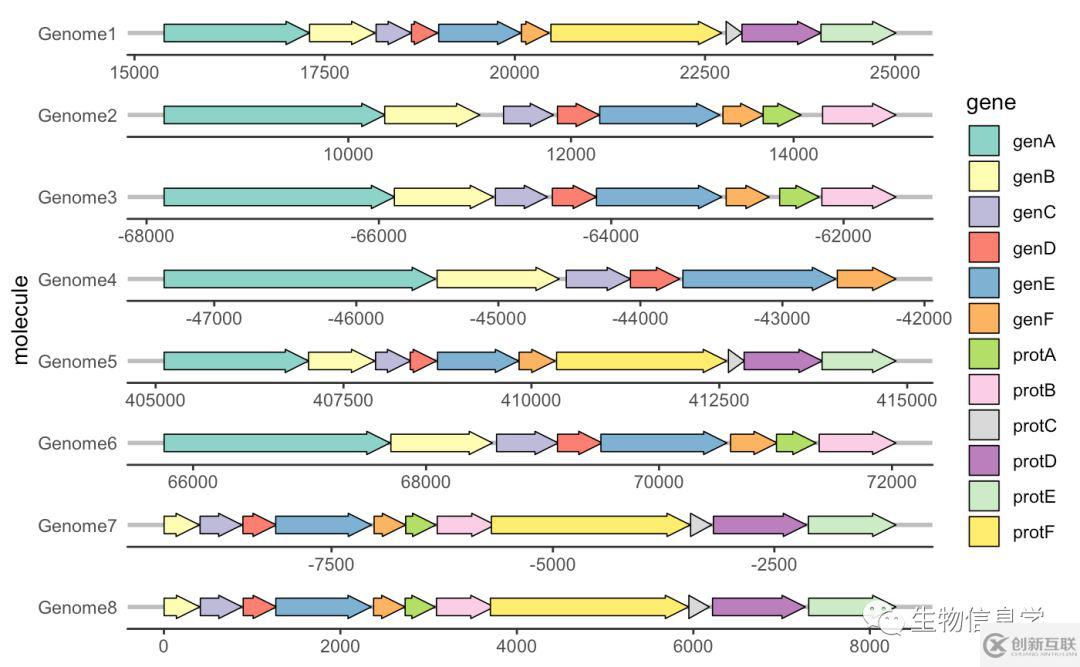
可以看出來,使用的是我們熟悉的ggplot2語法,再加上 geom_gene_arrow() 函數(shù)來實現(xiàn)了基因結(jié)構(gòu)的作圖。
下面的我們加上方向,也加上基因名稱,代碼如下:
ggplot(example_genes, aes(xmin = start, xmax = end, y = molecule, fill = gene, label = gene, forward = direction)) + geom_gene_arrow() + facet_wrap(~ molecule, scales = "free", ncol = 1) + scale_fill_brewer(palette = "Set3") + theme_genes() + geom_gene_label(align = "left")
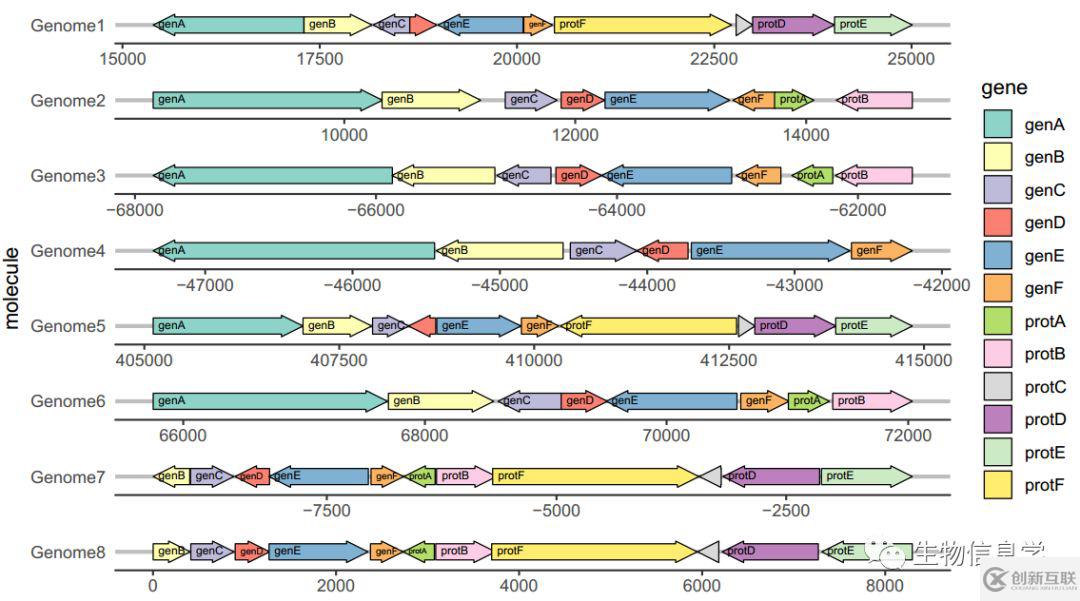
label = gene, forward = direction 指定了基因名和基因方向
geom_gene_label(align = "left") 在圖上添加了基因名并靠左對齊
3. 其他用法
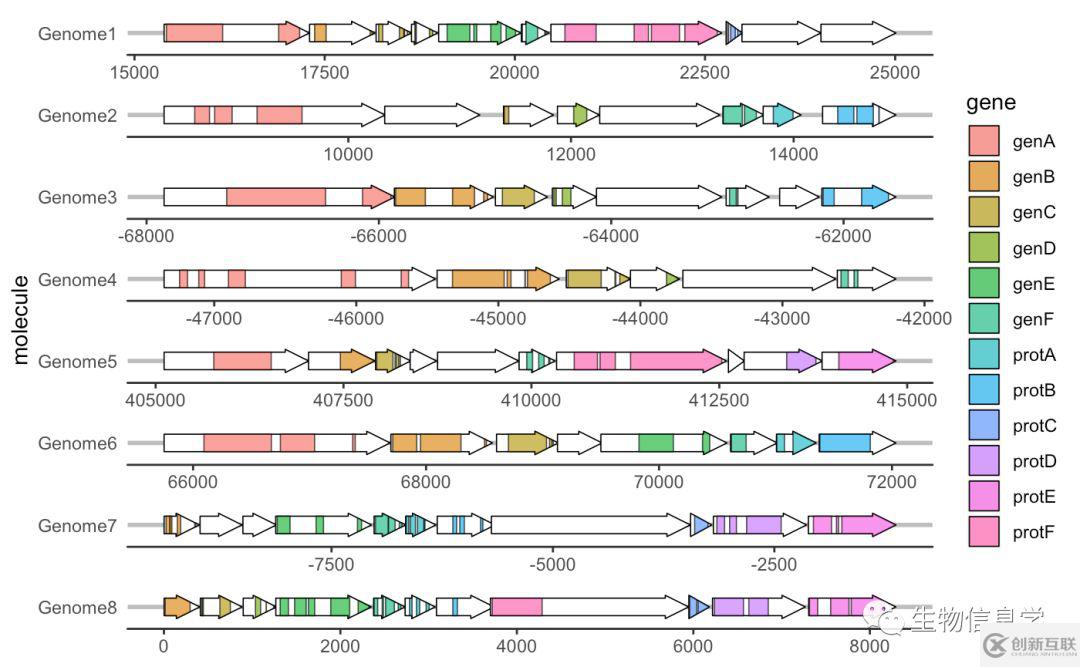
gggenes也可以如上圖所示展現(xiàn)基因結(jié)構(gòu)域特征或者比對信息,可以使用geom_subgene_arrow() 函數(shù)來實現(xiàn),需要額外用到另一份數(shù)據(jù)example_subgenes,代碼如下:
> head(example_subgenes) molecule gene start end strand subgene from to1 Genome5 genA 405113 407035 forward genA-1 405774 4065382 Genome5 genB 407035 407916 forward genB-1 407458 4078973 Genome5 genC 407927 408394 forward genC-1 407942 4081584 Genome5 genC 407927 408394 forward genC-2 408186 4082095 Genome5 genC 407927 408394 forward genC-3 408233 408257
ggplot(example_genes, aes(xmin = start, xmax = end, y = molecule)) + facet_wrap(~ molecule, scales = "free", ncol = 1) + geom_gene_arrow(fill = "white") + geom_subgene_arrow(data = example_subgenes, aes(xmin = start, xmax = end, y = molecule, fill = gene, xsubmin = from, xsubmax = to), color="black", alpha=.7) + theme_genes()
上述內(nèi)容就是如何用gggenes 畫基因結(jié)構(gòu)圖,你們學到知識或技能了嗎?如果還想學到更多技能或者豐富自己的知識儲備,歡迎關(guān)注創(chuàng)新互聯(lián)行業(yè)資訊頻道。
標題名稱:如何用gggenes畫基因結(jié)構(gòu)圖
標題路徑:http://vcdvsql.cn/article28/gjdgjp.html
成都網(wǎng)站建設公司_創(chuàng)新互聯(lián),為您提供建站公司、網(wǎng)頁設計公司、網(wǎng)站收錄、關(guān)鍵詞優(yōu)化、網(wǎng)站建設、搜索引擎優(yōu)化
聲明:本網(wǎng)站發(fā)布的內(nèi)容(圖片、視頻和文字)以用戶投稿、用戶轉(zhuǎn)載內(nèi)容為主,如果涉及侵權(quán)請盡快告知,我們將會在第一時間刪除。文章觀點不代表本網(wǎng)站立場,如需處理請聯(lián)系客服。電話:028-86922220;郵箱:631063699@qq.com。內(nèi)容未經(jīng)允許不得轉(zhuǎn)載,或轉(zhuǎn)載時需注明來源: 創(chuàng)新互聯(lián)

- 移動網(wǎng)站建設如何吸引用戶? 2023-01-30
- 成都網(wǎng)站建設:移動網(wǎng)站建設和微官網(wǎng)建設有哪些區(qū)別 2017-01-08
- 移動網(wǎng)站建設哪些方面要引起建站者重視? 2015-11-13
- 看完這7點,寫出有銷售力的電商文案 2022-06-10
- 移動網(wǎng)站建設和優(yōu)化需要注意的幾點 2021-09-16
- 移動網(wǎng)站建設需要注意哪些影響因素 2022-07-22
- 關(guān)于企業(yè)移動網(wǎng)站建設推廣計劃一點建議 2021-07-29
- 移動網(wǎng)站建設的好處 2016-11-08
- 移動網(wǎng)站建設是未來的一個大好市場 2020-11-07
- 什么是鄭州網(wǎng)站設計公司移動網(wǎng)站建設流程 2023-03-18
- 移動網(wǎng)站建設其實也有硬傷 怎樣做才能有效預防呢 2016-05-09
- 百度移動搜索:移動網(wǎng)站建設常見問題集錦 2022-02-22