怎么使用NetMHCpan進行腫瘤新抗原預測分析
本篇文章為大家展示了怎么使用NetMHCpan進行腫瘤新抗原預測分析,內容簡明扼要并且容易理解,絕對能使你眼前一亮,通過這篇文章的詳細介紹希望你能有所收獲。
10年積累的網站設計制作、成都做網站經驗,可以快速應對客戶對網站的新想法和需求。提供各種問題對應的解決方案。讓選擇我們的客戶得到更好、更有力的網絡服務。我雖然不認識你,你也不認識我。但先網站制作后付款的網站建設流程,更有高陽免費網站建設讓你可以放心的選擇與我們合作。
NetMHCpan軟件用于預測肽段與MHC I型分子的親和性,最新版本為v4.0, 基于人工神經網絡算法,以180000多個定量結合數據和MS衍生的MHC洗脫配體的組合為訓練集構建模型。結合親和力數據來自人,小鼠,豬等多個物種的MHC分子,MS洗脫的配體數據來自55個人和小鼠的HLA等位基因。
直接上傳fasta格式的蛋白序列就可以了, 示意如下
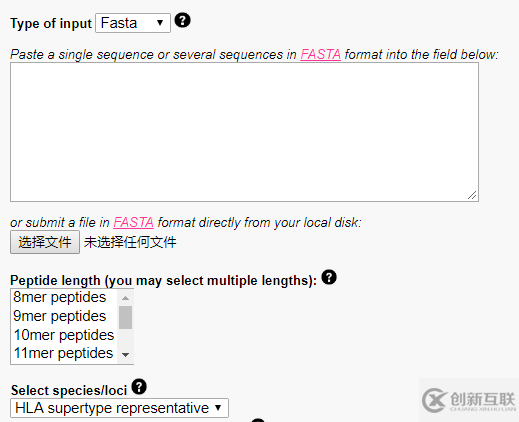
第一步上傳涵蓋了體細胞突變位點的氨基酸序列,上傳的氨基酸序列是突變之后的序列,不是野生型的蛋白質序列。
第二步選擇切割肽段的方式,抗原通過抗原表位與MHC分子結合,MHC I型分子可以結合的抗原表位長度為8到11個氨基酸,對應這里的8-11mer,先將蛋白質序列切分成短的肽段之后在進行MHC分子親和性的預測。
第三步選擇HLA allel, 確定之后點擊提交按鈕即可。輸出結果示意如下
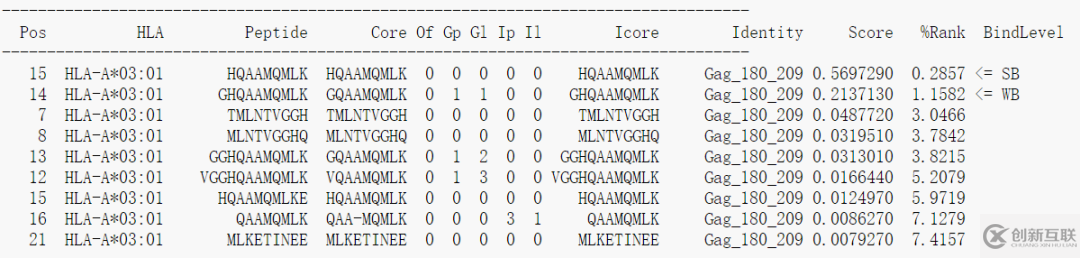
列數很多,其中的Peptide就是從原始的輸入序列中提取出的長度為8-11個氨基酸的肽段,Pos對應肽段的在原始序列上的起始位置,第一個位置從0開始計數;Core對應與MHC結合的肽段序列,和blast類似,允許插入和缺失,%Rank代表該肽段是一個天然存在的肽段的可能性,數值越小越好,最后一列的BindLevel代表親和力的強弱水平,SB表示strong binding, WB表示weak bingding。每一列的詳細解釋參見以下鏈接
http://www.cbs.dtu.dk/services/NetMHCpan/output.php
官方按照Rank值來篩選結果,默認情況下rank小于0.5的定義為強親和性,rank值在0.5到2之間的定義為弱親和性。通過該軟件可以從突變之后的氨基酸序列中預測到與MHC I型分子親和力較強的肽段,作為候選的腫瘤新抗原。
為了進一步簡化分析,相關的數據分析pipeline被開發出來,只需要提供腫瘤患者的體細胞突變數據和HLA分型結果即可,軟件自動提取突變氨基酸序列,并進行NetMHCpan分析,類似的軟件有很多,NeoPredPipe軟件就是其中之一,該軟件的網址如下
https://github.com/MathOnco/NeoPredPipe
基本用法如下
python NeoPredPipe.py \
-I somatic.vcf \
-H hlatypes.txt \
-o ./ \
-n TestRun \
-c 1 2 -E 8 9 10需要提供兩個輸入文件,-I指定體細胞突變的vcf文件,-H指定HLA分型結果文件。更多細節請參考該軟件的官方文檔。
通過上述的數據分析,可以快速定位出候選的新抗原,然而其中的假陽性率還是非常高的,后續還需要結合體外實驗來進一步篩選和過濾。
上述內容就是怎么使用NetMHCpan進行腫瘤新抗原預測分析,你們學到知識或技能了嗎?如果還想學到更多技能或者豐富自己的知識儲備,歡迎關注創新互聯行業資訊頻道。
當前文章:怎么使用NetMHCpan進行腫瘤新抗原預測分析
網頁地址:http://vcdvsql.cn/article28/gjihjp.html
成都網站建設公司_創新互聯,為您提供網站導航、用戶體驗、外貿網站建設、網頁設計公司、、服務器托管
聲明:本網站發布的內容(圖片、視頻和文字)以用戶投稿、用戶轉載內容為主,如果涉及侵權請盡快告知,我們將會在第一時間刪除。文章觀點不代表本網站立場,如需處理請聯系客服。電話:028-86922220;郵箱:631063699@qq.com。內容未經允許不得轉載,或轉載時需注明來源: 創新互聯

- google競價排名包年服務警剔 2016-05-29
- 怎樣解決Google有害信息的網站提示 2016-04-10
- Google正式宣布向移動優先索引轉移 2016-11-07
- 成都谷歌推廣商告訴你利用 Google Play 政策來打造用戶信任的應用 2016-04-09
- Google搜索排名怎么提升? 2015-01-12
- 外貿網站設計如何被Google快速收錄 2016-01-13
- 什么是Google SEO(谷歌優化)? 2023-05-05
- 分析Google Adwords和谷歌優化比較 2016-04-05
- 「關鍵字排名的規范」Google評定關鍵字排名的規范 2016-04-20
- 網站出現重復內容會對Google SEO優化有何影響? 2013-07-20
- 成都外貿推廣:充分利用新Google Search Console的7個步驟 2016-03-01
- google優化排名怎么做? 2016-09-26